ディープラーニングによるCovid-19X線画像分類器の説明可能性と可視性
キーワード: ディープラーニング、Grad-CAM、X線、COVID-19、HealthShare、IRIS
目的
イースターウィークエンド中に、Covid-19に感染した胸部X線画像分類とCT検出デモを実行するを触ってみました。 デモの結果は上出来で、このトピックに関するその頃の学術研究出版物に一致しているようでした。 でも、本当に「上出来」と言えるのでしょうか。
最近、「機械学習における説明可能性」に関するオンラインランチウェビナーを聴くことがあったのですが、たまたまその講演の最後でDonがこの分類結果について話していました。

上図は研究論文にも示されています。『“Why Should I Trust You?” Explaining the Predictions of Any Classifier』という論文です。 分類器は実際に、ペットの犬か野生の狼に分類するための主な入力として、雪といった自然環境などの背景ピクセルを取るようにトレーニングされていることがわかります。
これを見て、私は以前から持っていた関心を思い出しました。このやり方に、確かに好奇心を掻き立てられます。
- どのピクセルが実際に「Covid-19に感染した胸部」の結果に寄与しているかを知るには、一般的に「ブラックボックス」として提示されるこれらのCovid-19分類器をどのように「調べる」とよいのでしょうか。
- 最も単純な形式またはツールで、このケースに活用できる最も単純なアプローチは何でしょうか。
これは、過程を記録した10分程度のメモです。 最後に、なぜこのことが、今後の新しい刺激的なHealthShare機能に関係しているかについても触れるかもしれません。
範囲
幸いなことに、過去数年間において、さまざまなCNN派生の分類器用に便利なツールが登場しています。
- CAM(クラス活性化マップ): この応用は、こちらとこちらでよく説明されています。
- Grad-CAM(勾配重み付けクラス活性化): より汎用的なバージョンのCAMで、モデル全体内の任意のCNNレイヤーを調べることができます。
ここでは、Grad-CAMを使用して、前の記事で取り上げたCovid-19感染胸部画像分類器の簡単なデモを行います。
「Tensorflow 2.2.0rc + Jupyter」Dockerは、Nvidia T4 GPUを搭載したAWS Ubuntu 16.04サーバーで使用されます。 TensorFlow 2には、簡単な「勾配テープ」実装が用意されています。
以下は、Ubuntuサーバーで起動するための簡単な覚書です。
docker run -itd --runtime=nvidia -v /zhong/tf/:/tf -p 8896:8888 -p 6026:6006 --name tf-gpu2 tensorflow/tensorflow:2.2.0rc2-gpu-py3-jupyter
手法
上記のGrad-CAMに関する研究出版物から抜粋されている数学は無視しても構いません。
ここには、より透過的に結果を示せるよう、元の提案(ページ4、5)と後で使用されるPythonコードを継続的にクロスチェックできるように抜粋されています。


(1): 任意のクラスcの幅uと高さvのクラス識別的ローカリゼーションマップを取得するには、まず、畳み込みレイヤーの特徴マップAkに関してクラスc, yc(ソフトマックスの前)のスコアの勾配を計算します。 逆流しているこれらの勾配は、ターゲットクラスのニューロン重要度重みakを取得するためにグローバル平均プールされます。
(2): ターゲットクラスcのakを計算したら、重み付けされた活性化マップの組み合わせを実行し、ReLUで追跡します。 これにより、畳み込み特徴マップと同じサイズの粗いヒートマップが生成されます。
テスト
では、これまでに見つかった最も単純なコーディングを試してみましょう。
1. パッケージをインポートする
import tensorflow as tf;
print(tf.__version__)
2.2.0-rc2
import tensorflow as tf
import tensorflow.keras.backend as K
from tensorflow.keras.applications.inception_v3 import InceptionV3
from tensorflow.keras.preprocessing import image
from tensorflow.keras.applications.inception_v3 import preprocess_input, decode_predictions
import numpy as np
import os
import imutils
import matplotlib.pyplot as plt
import cv2
2. 以前にトレーニングして保存したモデルを読み込む
new_model = tf.keras.models.load_model('saved_model/inceptionV3')<br>new_model.summary()

モデル内の最後のCNNレイヤーの4Dは、最後のグローバルな平均プーリングが行われる前に「mixed10」と呼ばれていることがわかります。
3. Grad-CAMヒートマップを計算する
上記のGrad-CAMの式(1)および(2)を実装した、以下のような単純なバージョンのヒートマップがあります。 こちらの記事で説明されています。
with tf.GradientTape() as tape:
last_conv_layer = model.get_layer('mixed10')
iterate = tf.keras.models.Model([model.inputs], [model.output, last_conv_layer.output])
model_out, last_conv_layer = iterate(testX)
class_out = model_out[:, np.argmax(model_out[0])]
grads = tape.gradient(class_out, last_conv_layer)
pooled_grads = K.mean(grads, axis=(0, 1, 2)) heatmap = tf.reduce_mean(tf.multiply(pooled_grads, last_conv_layer), axis=-1)
この場合、NumPy配列 (27, 6, 6) のヒートマップが生成されます。 次に、そのサイズを元のX線画像のサイズに変更し、そのX線画像の上にオーバーレイします。これで完了です。
ただし、このケースでは、もう少し詳細なバージョンを使用します。これについては、こちらの記事で非常によく説明されています。 元のX線画像のサイズに変更されたGrad-CAMヒートマップで関数を構成しています。
# 必要なパッケージをtensorflow.keras.models
からインポート モデルをインポート
tensorflowをtfとしてインポート
numpyをnpとしてインポート
cv2をインポート
class GradCAM:
def __init__(self, model, classIdx, layerName=None):
self.model = model
self.classIdx = classIdx
self.layerName = layerName
if self.layerName is None:
self.layerName = self.find_target_layer()
def find_target_layer(self):
for layer in reversed(self.model.layers):
# このレイヤーに4D出力があるかをチェック
if len(layer.output_shape) == 4:
return layer.name raise ValueError("Could not find 4D layer. Cannot apply GradCAM.")
def compute_heatmap(self, image, eps=1e-8):br> gradModel = Model(
inputs=[self.model.inputs],
outputs=[self.model.get_layer(self.layerName).output,
self.model.output]) # 自動微分の演算を記録
with tf.GradientTape() as tape:
inputs = tf.cast(image, tf.float32)
(convOutputs, predictions) = gradModel(inputs)
loss = predictions[:, self.classIdx] # 自動微分を使って勾配を計算
grads = tape.gradient(loss, convOutputs) # guided gradientsを計算
castConvOutputs = tf.cast(convOutputs > 0, "float32")
castGrads = tf.cast(grads > 0, "float32")
guidedGrads = castConvOutputs * castGrads * grads convOutputs = convOutputs[0]
guidedGrads = guidedGrads[0] weights = tf.reduce_mean(guidedGrads, axis=(0, 1))
cam = tf.reduce_sum(tf.multiply(weights, convOutputs), axis=-1)
# ヒートマップのサイズを元のX線画像のサイズに変更する
(w, h) = (image.shape[2], image.shape[1])
heatmap = cv2.resize(cam.numpy(), (w, h))
# ヒートマップを正規化する
numer = heatmap - np.min(heatmap)
denom = (heatmap.max() - heatmap.min()) + eps
heatmap = numer / denom
heatmap = (heatmap * 255).astype("uint8")
# 生成されたヒートマップを呼び出し元関数に戻す
return heatmap
4. Covid-19に感染した胸部X線画像を読み込む
ここで、モデルのトレーニングと検証プロセスで使用されなかったテストX線画像を読み込みます。 (前の記事でもアップロードしたものです)
filename = './test/nejmoa2001191_f1-PA.jpeg'
orignal = cv2.imread(filename)
plt.imshow(orignal)
plt.show()

次に、256 x 256にサイズ変更し、0.0~1.0のピクセル値のNumPy配列「dataXG」に正規化します。
orig = cv2.cvtColor(orignal, cv2.COLOR_BGR2RGB)
resized = cv2.resize(orig, (256, 256))
dataXG = np.array(resized) / 255.0
dataXG = np.expand_dims(dataXG, axis=0)
**5. 簡易分類を実行する **
上記で新たに読み込まれたモデルを呼び出して、簡単な予測を実行することができます。
preds = new_model.predict(dataXG)
i = np.argmax(preds[0])
print(i, preds)
0 [[0.9171522 0.06534185 0.01750595]]
これは、0.9171522の確率でタイプ0、つまりCovid-19の胸部画像として分類されています。
6. Grad-CAMヒートマップを計算する
# ステップ3に基づいてヒートマップを計算する
cam = GradCAM(model=new_model, classIdx=i, layerName='mixed10') # このケースでは最後の4d形状「mixed10」を見つける
heatmap = cam.compute_heatmap(dataXG)
# 計算されたヒートマップを表示する
plt.imshow(heatmap)
plt.show()

7. ヒートマップを元のX線画像に表示する
# 上記と同様に透明なヒートマップを元の画像にオーバーレイする標準的な方法
heatmapY = cv2.resize(heatmap, (orig.shape[1], orig.shape[0]))
heatmapY = cv2.applyColorMap(heatmapY, cv2.COLORMAP_HOT) # COLORMAP_JET、COLORMAP_VIRIDIS、COLORMAP_HOT
imageY = cv2.addWeighted(heatmapY, 0.5, orignal, 1.0, 0)
print(heatmapY.shape, orig.shape)
# 元のX線画像、ヒートマップ、およびオーバーレイを合わせて描画する
output = np.hstack([orig, heatmapY, imageY])
fig, ax = plt.subplots(figsize=(20, 18))
ax.imshow(np.random.rand(1, 99), interpolation='nearest')
plt.imshow(output)
plt.show()
(842, 1090, 3) (842, 1090, 3)
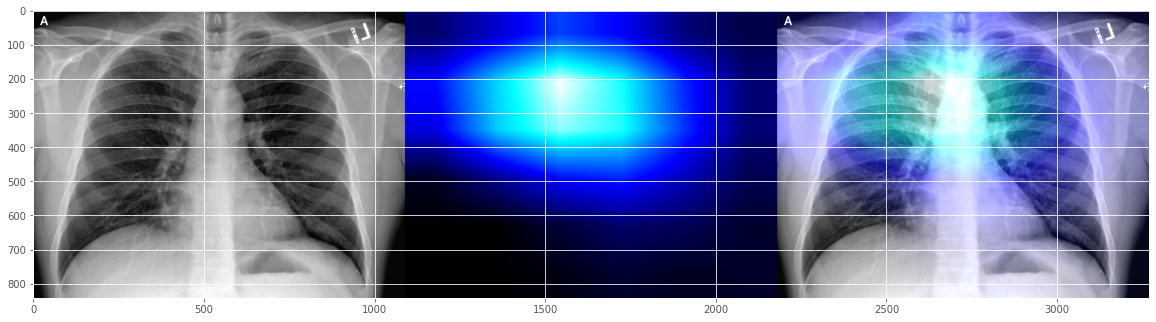 このCovid-19デモ分類器は、患者が「右傍気管線」周りに少し「混濁」の問題を抱えていると「信じている」ことを示すようです。 実際の放射線技師に確認を取らないと、私にはよくわかりません。
このCovid-19デモ分類器は、患者が「右傍気管線」周りに少し「混濁」の問題を抱えていると「信じている」ことを示すようです。 実際の放射線技師に確認を取らないと、私にはよくわかりません。
では、実際のケースからGitHubリポジトリに提出されたテスト画像をさらにいくつか試してみましょう。
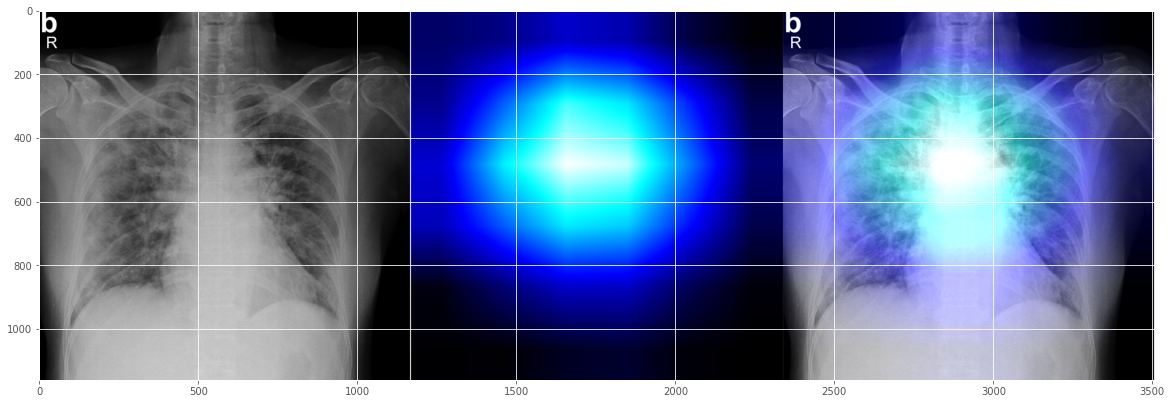
filename = './test/1-s2.0-S0929664620300449-gr2_lrg-b.jpg'
0 [[9.9799889e-01 3.8319459e-04 1.6178709e-03]]
これは、左の心臓ラインのエリア寄りで問題が起きていることが示されており、合理的に見えるCovid-19の解釈にも見えます。
別のランダムなテストX線画像を試してみましょう。
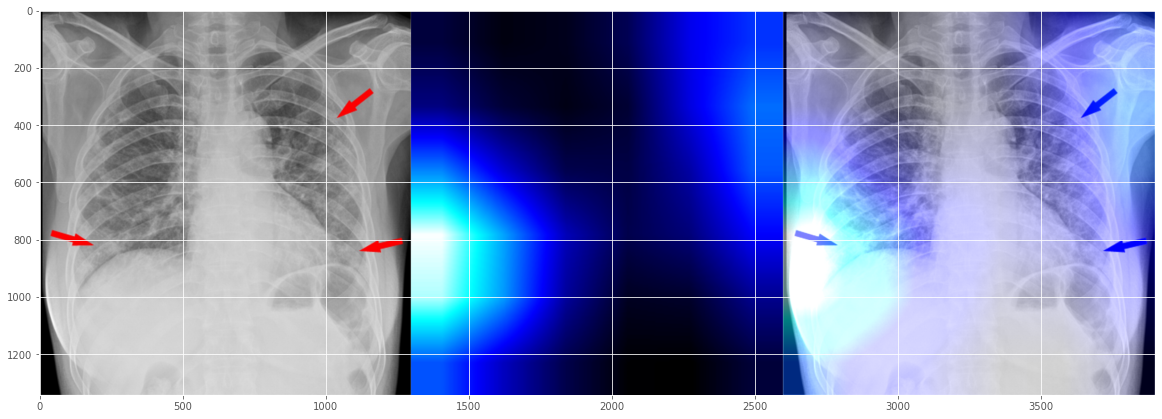
filename = '../Covid_M/all/test/covid/radiol.2020200490.fig3.jpeg'
0 [[0.9317619 0.0169084 0.05132957]]
驚いたことに、これは正確には見えませんが、もう一度見てみると、それほど不正確というわけでもないようですよね。 問題のエリアが2つ示されています。左側の大きな問題と右側のある程度の問題です。人間の放射線技師がマークアップしたものとある程度一致しているようですね (人間のマーカーに対してトレーニングされていないことを願っていますが。そうなれば、別の説明可能性レベルの問題になってしまいますから)。
この10分間の簡易メモでX線画像の読み取りに興味がある人はほとんどいないでしょうから、ここでストップすることにします。
理由
個人的に、「説明可能性」と「解釈可能性」の重要性、そしてそれらへの技術的アプローチを深く重んじています。 この次元にわずかでも試みることは、どんなに小さくとも努力する価値があります。 最終的には「データの公平性」、「データの正義」、および「データの信頼性」はデジタル経済のプロセスの透明性に基づいて構築されるようになるでしょうし、 現在でも利用可能になり始めています。 1995年の夏、私がまだ若く博士論文を書いていた25年前には、主にブラックボックスとして使用されていたいわゆる「ニューラルネットワーク」を理解しようとさえ思ってもいませんでした。 当時AIは論理的推論マシンとしての「エキスパートシステム」と考えられており、「ニューラルネットワーク」は「ニューラルネットワーク」であり、「ディープラーニング」は生まれてもいませんでした。 現在では、今日のAI開発者がすぐに利用できる研究やツールがますます増えています。
最後になりましたが、このデモに限定してこのようなツールについて評価できることは、最初からピクセルグレードのラベリングである必要がないということです。胸部の病変領域を自動的に生成しようとしたことから、これはある種の半自動ラベリングです。 実際の作業に大きな意味を持っています。 昨年、放射線技師の友人が、骨折データのU-Netトレーニングでピクセルグレードのラベルを幾度となく生成するのを手助けしてくれたのを覚えています。本当に目が痛くなる作業でした。
今後の内容
少しさまよってしまいました。 過去10年以上においてディープラーニングが急進したお陰で、医用画像はAI分野で比較的成熟した方向性となっています。 良い安定した時期を楽しむ価値があるでしょう。 ただし次は、少し時間があるのであれば、NLPの面により取り組んでみたいと思います。
謝辞
すべての出典は必要に応じて上記の文中に示されています。 必要であれば、さらに参考資料を追加したいと思います。
免責事項:
繰り返しになりますが、上記は、今記録しておかなければまた数週間が過ぎてしまう可能性に備えた簡単なメモです。 すべては「開発者」としての個人的見解です。 この内容は、必要に応じていつでも変更されます。 上記は、臨床的解釈と言うより、技術的なアイデアとアプローチを紹介することを目的としています。臨床的解釈については、十分なデータの質と量に基づくゴールデンルールをセットアップするには、専門の放射線技師が必要となるでしょう。