Covid-19に感染した胸部X線画像分類とCT検出デモを実行する
キーワード: COVID-19、医用画像、ディープラーニング、PACSビューア、HealthShare。
目的
私たちは皆、この前例のないCovid-19パンデミックに悩まされています。 現場のお客様をあらゆる手段でサポートする一方で、今日のAI技術を活用して、Covid-19に立ち向かうさまざまな前線も見てきました。
昨年、私はディープラーニングのデモ環境について少し触れたことがあります。 この長いイースターの週末中に、実際の画像を扱ってみてはどうでしょうか。Covid-19に感染した胸部X線画像データセットに対して簡単な分類を行うディープラーニングモデルをテスト実行し、迅速な「AIトリアージ」や「放射線科医の支援」の目的で、X線画像やCT用のツールがdockerなどを介してクラウドにどれほど素早くデプロイされるのかを確認してみましょう。
これは、10分程度の簡易メモです。学習過程において、最も単純なアプローチでハンズオン経験を得られることを願っています。
範囲
このデモ環境では、次のコンポーネントが使用されます。 これまで見てきた中で最も単純な形式です。
- 3タイプの小さな匿名加工オープンデータセット: Covid-19感染胸部、細菌性肺炎胸部、正常な胸部。
- 1セットのディープラーニングモデル(X線胸部画像分類用のInception V3モデル)
- JupyterノートブックによるTensorFlow 1.13.2コンテナ
- GPU用のNvidia-Docker2コンテナ
- Nvidia T4 GPU搭載AWS Ubuntu 16.04 VM(事前トレーニング済みモデルの再トレーニングを行わない場合はノートパソコンのGPUで十分です)
および
- 「AI支援CT検出」のデモコンテナ
- サードパーティのOpen PACS Viewerのデモコンテナ
- HealthShare Clinical Viewerのデモインスタンス
以下は、このデモの範囲では説明されていません。
- PyTorchの人気が高まっています(次の機会に利用します)
- デモ環境では、TensorFlow 2.0の実行速度がはるかに低下します(そのためバージョン1.13に戻します)
- AutoMLなどのマルチモデルアンサンブル(実際に人気が高まっていますが、この小さなデータセットでは昔ながらのシングルモデルで十分です)
- 実際のサイトから得るX線およびCTデータ。
免責事項
このデモは、この特定の分野における臨床試験についてではなく、技術的なアプローチに関するものです。 CTおよびX線などの証拠に基づくCovid-19診断は現在ではオンラインで広く入手可能であり、肯定的なレビューもあれば、否定的なレビューもあります。また、このパンデミックにおいて、国や文化における役割が実際に異なります。 また、この記事のコンテンツとレイアウトは、必要に応じて変更される場合があります。 このコンテンツは「開発者」としての純粋な個人的見解です。
データ
このテストの元の画像は、Joseph Paul Cohenによって一般公開されているCovid-19 Lung X-Ray setに含まれるものと、オープン形式のKaggle Chest X-Ray setsからAdrian YuがGradientCrescentリポジトリの小さなテストセットに集めたの正常な胸部画像です。 また、皆さんが使用できるように、簡単なテスト用のテストデータをこちらにアップロードしました。 これまでのところ、以下の小さなトレーニングセットが含まれます。
- Covid-19に感染した胸部画像 60点
- 正常な胸部画像 70点
- 細菌性肺炎に感染した胸部画像 70点
テスト
以下のテストでは、独自のフレーバーに合わせてわずかに変更したものを実行しました。
- ベースのInception V3モデルとその上の2つのCNNレイヤー
- 再トレーニング用に基盤のInceptionレイヤーの非凍結重みを使った転移学習(ノートパソコンのGPUでは、事前トレーニング済みのInceptionレイヤーを凍結するだけです)
- これまでに収集した小さなデータセットを補うためのわずかなデータ拡張
- バイナリの代わりに3つのカテゴリを使用: Covid-19、正常、細菌性(またはウイルス性)肺炎(これらの3つのクラスを使用した理由は後で説明します)
- 後のステップで使えるテンプレートとして、基本的な3クラス混同行列を計算
注意: ほかの一般的なCNNベースのモデル(VGG16やResNet50など)ではなく、Inception V3を選択した理由は特にありません。 他にもモデルがありますが、最近、骨折データセットのデモ実行に使用したため、ここではそれを再利用しているだけです。 以下のJupyterノートブックスクリプトを実行し直す際には、お好きなモデルを使用してください。
この記事には、Jupyterノートブックも添付しました。 以下に簡単な説明も示しています。
1. 必要なライブラリをインポートする
# import the necessary packages
from tensorflow.keras.layers import AveragePooling2D, Dropout, Flatten, Dense, Input
from tensorflow.keras.models import Model
from tensorflow.keras.optimizers import Adam
from tensorflow.keras.utils import to_categorical
from tensorflow.keras import optimizers, models, layers
from tensorflow.keras.applications.inception_v3 import InceptionV3
from tensorflow.keras.applications.resnet50 import ResNet50
from tensorflow.keras.preprocessing.image import ImageDataGenerator
from sklearn.preprocessing import LabelEncoder, OneHotEncoder
from sklearn.model_selection import train_test_split
from sklearn.metrics import classification_report, confusion_matrix
from imutils import paths
import matplotlib.pyplot as plt
import numpy as np
import cv2
import os2. ここに提供されているサンプルの画像ファイルを読み込む
# set learning rate, epochs and batch size
INIT_LR = 1e-5 # この値は選択したモデルに固有の値です: Inception、VGG、ResNetなど。
EPOCHS = 50
BS = 8
print("Loading images...")
imagePath = "./Covid_M/all/train" # 同じ画像のローカルパスに変更してください
imagePaths = list(paths.list_images(imagePath))
data = []
labels = []
# 指定されたパスにあるすべてのX線画像を読み取り、サイズを256x256に変更します
for imagePath in imagePaths:
label = imagePath.split(os.path.sep)[-2]
image = cv2.imread(imagePath)
image = cv2.cvtColor(image, cv2.COLOR_BGR2RGB)
image = cv2.resize(image, (256, 256))
data.append(image)
labels.append(label)
#ピクセル値を0.0~1.0の実数に正規化します
data = np.array(data) / 255.0
labels = np.array(labels)
# マルチクラスのラベル付けを行うためにワンホットエンコーディングを実行します
label_encoder = LabelEncoder()
integer_encoded = label_encoder.fit_transform(labels)
labels = to_categorical(integer_encoded)
print("... ... ", len(data), "images loaded in multiple classes:")
print(label_encoder.classes_)Loading images... ... ... 200 images loaded in 3x classes: ['covid' 'normal' 'pneumonia_bac']
3. 基本的なデータ拡張を追加し、モデルを再構成してトレーニングする
# データとトレーニング用と検証用に分割します。 (trainX, testX, trainY, testY) = train_test_split(data, labels, test_size=0.20, stratify=labels, random_state=42) # 単純な拡張を追加します。 注意: このケースでは、拡張が多すぎるとうまく行きません。テストの過程でわかりました。 trainAug = ImageDataGenerator(rotation_range=15, fill_mode="nearest") #事前トレーニング済み「ImageNet」の重みの転移学習でInceptionV3モデルを使用します。 #注意: VGG16またはResNetを選択した場合は、最上部で初期の学習率をリセットする必要がある場合があります。 baseModel = InceptionV3(weights="imagenet", include_top=False, input_tensor=Input(shape=(256, 256, 3))) #baseModel = VGG16(weights="imagenet", include_top=False, input_tensor=Input(shape=(256, 256, 3))) #baseModel = ResNet50(weights="imagenet", include_top=False, input_tensor=Input(shape=(256, 256, 3))) #Inception V3モデルの上にカスタムCNNレイヤーを2つ追加します。 headModel = baseModel.output headModel = AveragePooling2D(pool_size=(4, 4))(headModel) headModel = Flatten(name="flatten")(headModel) headModel = Dense(64, activation="relu")(headModel) headModel = Dropout(0.5)(headModel) headModel = Dense(3, activation="softmax")(headModel) # 最終モデルを構成します model = Model(inputs=baseModel.input, outputs=headModel) # Navidia T4 GPUを使用しているので、再トレーニングを行うために、事前トレーニング済みのInception「ImageNet」重みの凍結を解除します #baseModel.layersのレイヤーの場合: # layer.trainable = False print("Compiling model...") opt = Adam(lr=INIT_LR, decay=INIT_LR / EPOCHS) model.compile(loss="categorical_crossentropy", optimizer=opt, metrics=["accuracy"]) # 上記で事前トレーニング済みの重みの凍結を解除したため、全モデルをトレーニングします。 print("Training the full stack model...") H = model.fit_generator( trainAug.flow(trainX, trainY, batch_size=BS), steps_per_epoch=len(trainX) // BS, validation_data=(testX, testY), validation_steps=len(testX) // BS, epochs=EPOCHS)
... ... Compiling model... Training the full stack model... ... ... Use tf.cast instead. Epoch 1/50 40/40 [==============================] - 1s 33ms/sample - loss: 1.1898 - acc: 0.3000 20/20 [==============================] - 16s 800ms/step - loss: 1.1971 - acc: 0.3812 - val_loss: 1.1898 - val_acc: 0.3000 Epoch 2/50 40/40 [==============================] - 0s 6ms/sample - loss: 1.1483 - acc: 0.3750 20/20 [==============================] - 3s 143ms/step - loss: 1.0693 - acc: 0.4688 - val_loss: 1.1483 - val_acc: 0.3750 Epoch 3/50 ... ... ... ... Epoch 49/50 40/40 [==============================] - 0s 5ms/sample - loss: 0.1020 - acc: 0.9500 20/20 [==============================] - 3s 148ms/step - loss: 0.0680 - acc: 0.9875 - val_loss: 0.1020 - val_acc: 0.9500 Epoch 50/50 40/40 [==============================] - 0s 6ms/sample - loss: 0.0892 - acc: 0.9750 20/20 [==============================] - 3s 148ms/step - loss: 0.0751 - acc: 0.9812 - val_loss: 0.0892 - val_acc: 0.9750
4. 検証結果用に混同行列をプロットする
print("Evaluating the trained model ...")
predIdxs = model.predict(testX, batch_size=BS)
predIdxs = np.argmax(predIdxs, axis=1)
print(classification_report(testY.argmax(axis=1), predIdxs, target_names=label_encoder.classes_))
# 基本的な混同行列を計算します
cm = confusion_matrix(testY.argmax(axis=1), predIdxs)
total = sum(sum(cm))
acc = (cm[0, 0] + cm[1, 1] + cm[2, 2]) / total
sensitivity = cm[0, 0] / (cm[0, 0] + cm[0, 1] + cm[0, 2])
specificity = (cm[1, 1] + cm[1, 2] + cm[2, 1] + cm[2, 2]) / (cm[1, 0] + cm[1, 1] + cm[1, 2] + cm[2, 0] + cm[2, 1] + cm[2, 2])
# 混同行列、精度、感度、特異度を示します
print(cm)
print("acc: {:.4f}".format(acc))
print("sensitivity: {:.4f}".format(sensitivity))
print("specificity: {:.4f}".format(specificity))
# トレーニング損失と精度をプロットします
N = EPOCHS
plt.style.use("ggplot")
plt.figure()
plt.plot(np.arange(0, N), H.history["loss"], label="train_loss")
plt.plot(np.arange(0, N), H.history["val_loss"], label="val_loss")
plt.plot(np.arange(0, N), H.history["acc"], label="train_acc")
plt.plot(np.arange(0, N), H.history["val_acc"], label="val_acc")
plt.title("Training Loss and Accuracy on COVID-19 Dataset")
plt.xlabel("Epoch #")
plt.ylabel("Loss/Accuracy")
plt.legend(loc="lower left")
plt.savefig("./Covid19/s-class-plot.png")
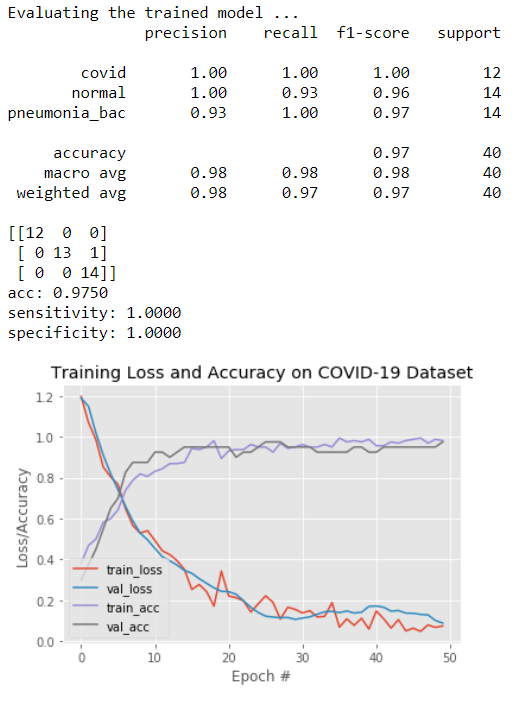
上の図から、小さなデータセットと5分未満の簡易トレーニングでも「転移学習」のメリットにより、結果はそれほど悪くないことがわかります。12点すべてのCovid-19に感染した胸部画像は正しく分類されており、誤って「細菌性肺炎」の胸部画像として分類された正常な胸部画像は、合計40点のうち1点のみです。
5. 実際のX線画像をテストするための混同行列をプロットする
では、さらにもう一歩飛躍してみましょう。簡単にトレーニングされたこの分類器がどれほど効果的であるかをテストするために、実際のX線画像を送信してみます。
上記のトレーニングや検証セットで使用していない27点のX線画像をモデルにアップロードしました。
Covid-19に感染した胸部画像 9点と正常な胸部画像 9点と細菌性肺炎に感染した胸部画像 9点です。 (これらの画像は、この記事にも添付されています。)
ステップ2のコードを1行だけ変更し、テスト画像が確実に異なるパスから読み込まれるようにしました。
...
imagePathTest = "./Covid_M/all/test"
...次に上記のトレーニング済みのモデルを使用して予測します。
predTest = model.predict(dataTest, batch_size=BS) print(predTest) predClasses = predTest.argmax(axis=-1) print(predClasses) ...
最後に、ステップ5のようにして、混同行列を再計算します。
testX = dataTest testY = labelsTest ... ...
実際のテスト結果が得られました。
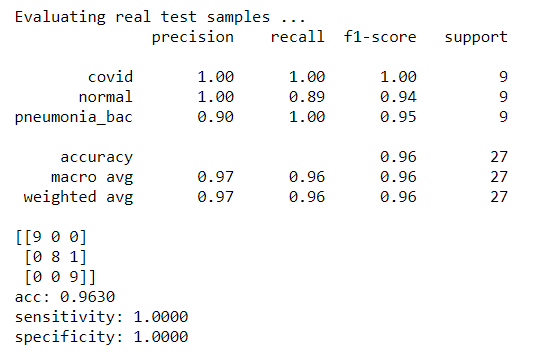
またしても、トレーニング済みモデルは、すべてのCovid-19に感染した胸部画像を正しく分類できるようです。 こんなに小さなデータセットにしては、それほど悪くない出来です。
6. さらなる監察結果
このイースターの週末に、さまざまなデータセットを試し、Covid-19に感染した胸部画像には明確な特徴があるように見えました。AI分類器の観点では、通常の細菌性またはウイルス性(インフルエンザ)感染症の影響を受けた胸部画像と区別することは比較的簡単です。
また、いくつかの簡単なテストから、細菌性とウイルス性(一般的なインフルエンザ)の胸部画像を区別するのは非常に困難であることに気づきました。 時間があれば、ほかのKaggle挑戦者がおそらくあの状況で行うように、クラスタアンサンブルを使用して、その違いを追跡しなければならないでしょう。
では、臨床の観点では、上記の結果は本当に正しいのでしょうか? Covid-19に感染した胸部にはX線で本当に明確な特徴が見られるのでしょうか? あまり確信が持てません。 実際の胸部放射線科医に意見を聞かなくてはいけないでしょう。 今のところは、現在のデータセットは結論を出すには小さすぎると思います。
次: 実際のサイトのX線画像をもっと集めて、xgsboot、AutoML、または新しいIRIS IntegratedMLワークベンチを使って、深く調べたいと思います。 さらに、医師やA&Eトリアージ向けに、Covid-19に感染した胸部画像を、その深刻度に応じてレベル1、レベル2、およびレベル3のようにさらに分類できるはずです。
とにかく話を戻すと、データセットと上記のJupyterノートブックを添付しました。
デプロイメント
上記では、この「医用画像」分野における簡単なセットアップについて、わりと単純な出発点に触れました。 このCovid-19フロントは、実際には、過去1年の週末や長期休暇中に調べたもののうち、3つ目の試行となります。 他には、「AI支援骨折検出」システムや「AI支援眼科網膜診断」システムなどを調べました。
上記のモデルは、今のところは単純すぎて試すほどでもないかもしれませんが、一般的な疑問はすぐにでも避けて通れないでしょう。ある種の「AIサービス」はどのようにデプロイすればよいのでしょうか?
テクノロジースタックとサービスのライフサイクルに関わることですが、どのような問題を解決しようとしているのか、どういった実際の価値が得られるのかといった実際の「ユースケース」によっても異なります。 答えはテクノロジーほど明確ではないこともあります。
イギリスのRCR(王立放射線学者会)の原案では、「放射線技師のAI支援」と「A&EまたはプライマリケアにおけるAIトリアージ」という2つの単純なユースケースが提案されました。 正直なところ現時点では2つ目の「AIトリアージ」に同意しており、個人的には、この方がより高い価値をもたらすのではないかと思っています。 また、幸いにも、今日の開発者はクラウド、Docker、AI、そしてもちろんInterSystemsのHealthShareにより、これまで以上の力を使ってこの種のケースを解決することができます。
たとえば、以下のスクリーンキャプチャは実際にAWSにホストされたエンタープライズ級の「AI支援によるCovid-19感染胸部のCT検出」サービスを示しており、デモの目的で、いかにしてこのサービスを直接HealthShare Clinical Viewerに組み込むかを説明しています。 X線撮影と同様に、DICOMに設定されたCTも迅速な「AIトリアージ」のユースケース用に毎日24時間稼働しながら、このオープンPACS Viewerに直接アップロードまたは送信し、「AI診断」をクリックするだけで、トレーニング済みモデルに基づいて定量化されたCovid-19感染確率の判定を提供することができます。 X線画像分類などのモデルは、最前線に立つ医師を支援できるよう、同一患者のコンテキストで既存のPACSビューアの上にデプロイし、同じ方法で呼び出すことができます。

謝辞
繰り返しますが、このテスト画像は、Joseph Paul Cohenによる Covid-19 Lung X-Ray setから得たもので、正常な胸部画像はAndrian YuがGradientCrescentリポジトリに収集しているオープンのKaggle Chest X-Ray setsに含まれるものです。 また、「テスト」セクションにリストされたとおり、独自に改善したトレーニングを使ってPyImageSearchのAdrianの構造を再利用しています。 また、テストデータベースを確認するために、X線画像とCT画像用のAIモジュール付きAWSクラウドベースOpen PACS Viewerを提供していただいたHYMにも感謝しています。
次の内容
今日、AIは、人間の健康と日常生活のほぼすべての側面を「浸食」しています。 単純化し過ぎた私の見解では、ヘルスケア分野におけるAI技術の応用には、以下のようないくつかの方向性があると考えられます。
- 医用画像: 胸部、心臓、目、脳などのX線、CT、またはMRIなどの診断画像。
- NLPによる読解: 膨大な量のテキスト資産とナレッジベースのマイニング、理解、学習。
- 公衆衛生: 疫学などの傾向予測、分析、モデリングなど。
- パーソナル化AI: 個人専用の健康アシスタントとして、ともに成長して老化するように特別にトレーニングされたAI/ML/DLモデル。
- その他のAI: AlphaGoや、Covid-19への対抗に活用できる3次元タンパク質構造予測向けのAlphaFoldなど。このような最先端の画期的な技術に非常に感激しています。
学習の過程で、焦点を当てられるものを検討していきたいと思います。 いずれにしても、在宅が長く続かない限りは、取り上げたいリストにしかすぎないかもしれません。
付録 - ファイルアップロードはこちらです。 上記で使用した画像と上記のJupyterノートブックファイルが含まれています。 週末に新規でセットアップして実行するには、数時間かかるかもしれません。